近日,厦门大学信息学院俞容山教授团队在cell 子刊patterns上发表题为 “daism-dnnxmbd: highly accurate cell type proportion estimation with in silico data augmentation and deep neural networks” 的研究工作。
自人类基因组计划以来,越来越强大的对基因、蛋白质和代谢物进行深度分子表征的工具不断问世。临床组织样本的分子谱分析是精准医学的核心,通过对比来自患者和对照组的组织样本,可以发现潜在的生物标志物并深入了解治疗效果。然而,组织微环境是细胞类型的复杂混合物。深入剖析免疫微环境、阐明混合细胞类型的贡献,并检测细胞群响应感染或药物的变化是极具挑战性的工作。
为了探究复杂组织微环境中的细胞组成及丰度,计算生物学家开发了多种反卷积算法,通过组织混合分子谱反卷积出样本的不同细胞比例。传统算法比如cibersort或xcell假定每种细胞类型都有特定的分子表达谱,混合分子谱是每种细胞类型特定表达的线性组合。因此,这些算法在预定义好的不同细胞类型的特征分子谱的基础上,通过最小二乘回归或者富集来获得细胞比例。然而,从生物学角度来说,与静态分子特征相比,细胞分子图谱是一个连续体,取决于细胞状态、生物条件和细胞类型,而不是一组离散的图谱。同时,每种细胞类型对复杂混合物的贡献是线性的这一假设并不现实。因此,这些基于预定义分子谱进行回归的方法存在一定的性能瓶颈。针对这个问题,人们进一步提出了基于深度学习(dnn)的细胞反卷积方法,例如scaden,使得从数据中公正地了解分子谱及其非线性组合成为可能。但是,dnn 方法需要大量的训练数据。虽然 scaden 利用单细胞数据构造训练数据的办法解决了这个问题。然而,单细胞数据构建的训练数据并不能保证与实际数据的统计分布相匹配。因此,这些算法很难在广泛的临床条件、测序平台、批次效应、实验条件和样本类型中获得准确的细胞比例结果。
针对这个难题,俞容山团队开发出一种数据驱动的细胞反卷积方法daism-dnnxmbd (图1)。该方法提出了干湿结合的策略,先通过湿实验的方法,获得特定实验条件下一定数量的校准样本,然后,再通过计算机混合策略的新型数据增强方法daism,从校准样本扩增出足够的训练数据进行深度神经网络(dnn)训练,从而获得针对目标实验条件的细胞比例预测反卷积模型。实验结果表明,对于现实世界数据集中评估的所有细胞类型,daism-dnn 预测的比例准确度始终优于其他现有方法(图2)。因此,通过严格的实验操作流程(sop),我们可以为多个生物医学实验创建“一次训练,多次重复使用”的daism-dnn模型,有望在大规模的临床研究应用中发挥重要的作用。
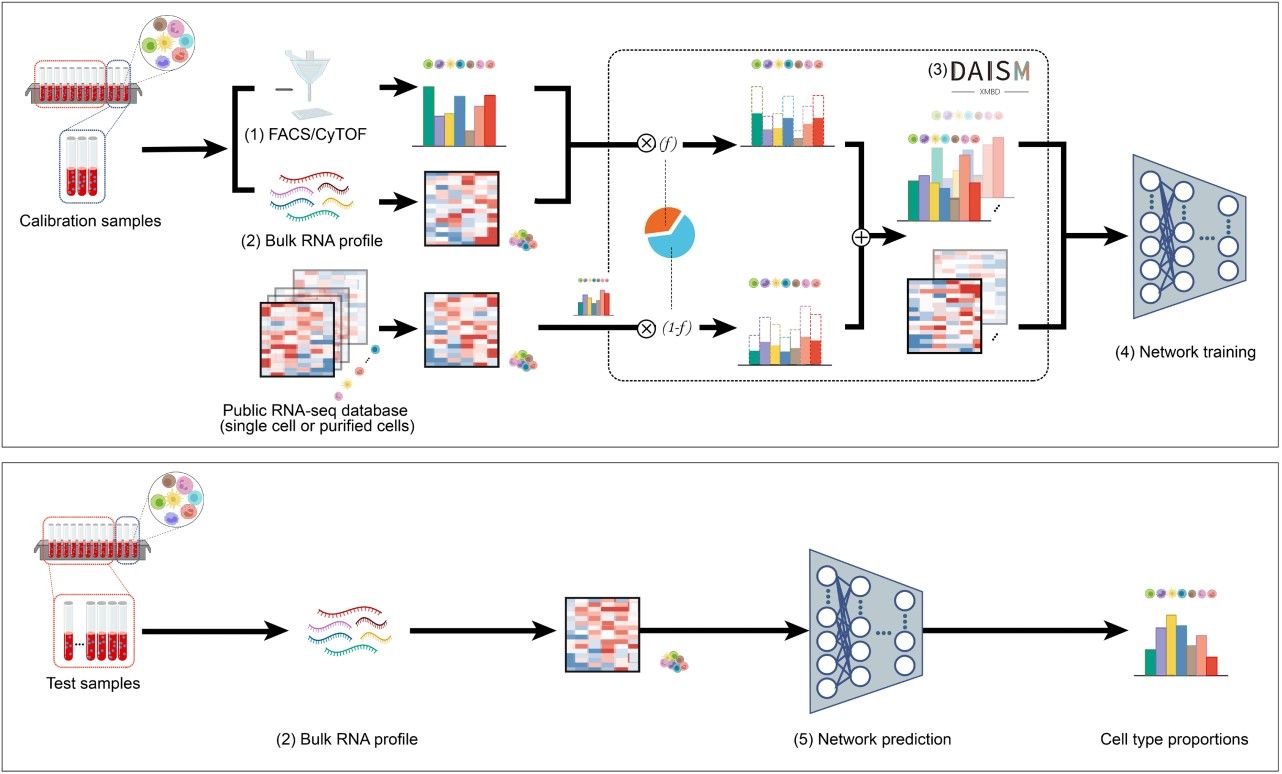
图1. daism-dnnxmbd工作流程. 来源:patterns
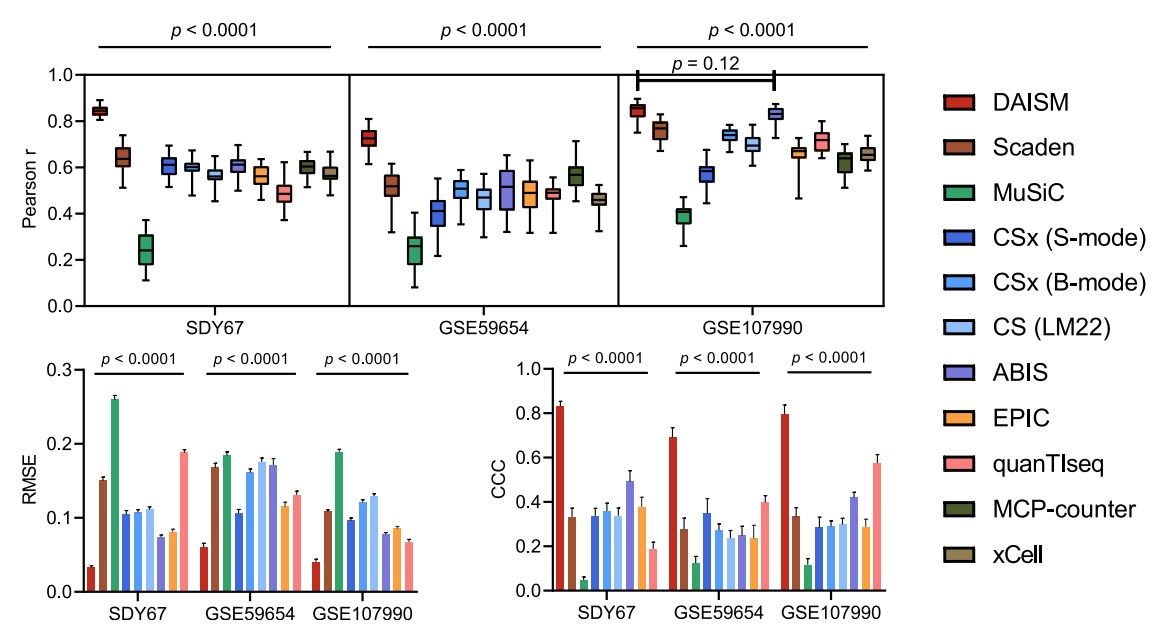
图2. daism-dnnxmbd和其他算法的性能评估与比较。来源:patterns
近日,trends in cell biology(if:20.808)以聚焦评测(spotlight)的形式 (“data-driven bioinformatics to disentangle cells within a tissue microenvironment”),高度评价了这项工作。该评测文章认为 daism 巧妙解决了数据驱动的机器学习在生命科学应用上难以做到的尊重个体实验参数和病理条件差异的难题,从而大幅度提高了反卷积算法的性能。该方法未来可望在包含 dna 甲基化或染色质可及性数据的生物数据处理上得到进一步应用。文章进一步认为目前机器学习在细胞成分分析上的应用还是太过依赖有监督学习的方法,未来,在缺乏有效病理标注的情况下,无监督学习应该会得到进一步的广泛应用(图3)。
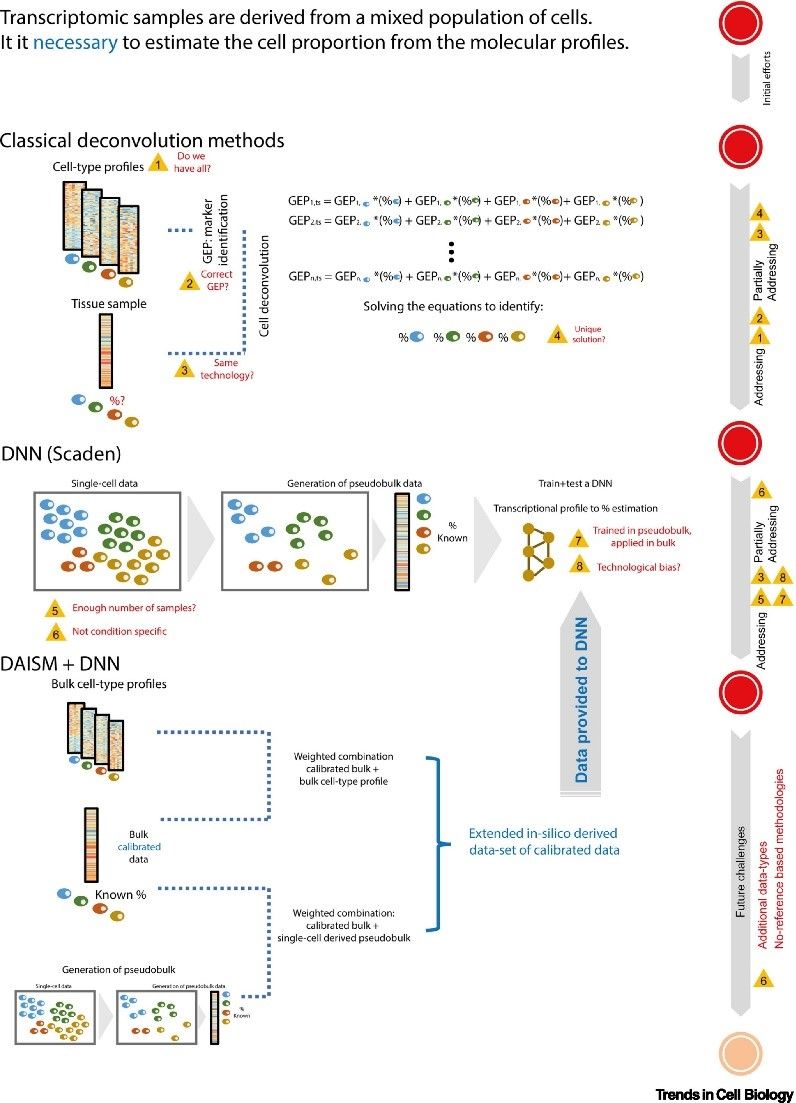
图3. 机器学习方法在细胞成分分析上应用的演进。来源:trends in cell biology
该项工作在俞容山教授的指导下完成,由信息学院博士生林雅婷,研究生李豪钧完成主要研究工作,为文章的共同第一作者。厦门大学信息学院俞容山教授,厦门大学健康医疗大数据国家研究院韩家淮院士,厦门极元科技有限公司杨文娴博士为文章共同通讯作者。厦门大学生命科学学院张蕾老师,厦门大学医学院王科嘉教授,厦门艾德生物医药科技股份有限公司也对本研究工作给予大力帮助和支持。
该研究工作的一个早期模型还参加了国际计算生物医学领域最具影响力之一的算法挑战赛——dream challenge tumor deconvolution比赛。团队提交的细胞丰度估计模型取得了优于所有其他团队提交的模型的性能,勇夺第一。
参考资料:
[1] tegner j n, gomez-cabrero d. data-driven bioinformatics to disentangle cells within a tissue microenvironment[j]. trends in cell biology, 2022.
[2] lin y, li h, xiao x, et al. daism-dnnxmbd: highly accurate cell type proportion estimation with in silico data augmentation and deep neural networks[j]. patterns, 2022, 3(3): 100440.